Desafio Marzo/Abril ADN Forense
ACTUALIZACIÓN 1 DE MAYO 21:00 horas Estimados amigos, el plazo para participar ya se cumplió, tenemos 6 participantes. Lamentablemente no he podido organizar el tiempo para revisar las participaciones. Así que los resultados estarán dentro de los próximos días. Saludos y gracias a todos los que han participado.
Uno de los problemas comunes en bio informática es el de tratar de encontrar similitud entre secuencias de genes.
Los genes, en bio informática, se representan como secuencias de 4 posibles letras A,C, G o T (A de Adenina, C de Citosina, G de Guanina y T de Timina, las cuatro bases de los ácidos nucléicos como el ADN o el ARN).
Una manera de determinar la similitud de dos secuencias de ADN es usando una técnica conocida como alineamiento de secuencias.
El proceso de alineamiento de secuencias de genes es como sigue.
Dados dos genes, por ejemplo: AGTGATG y GTTAG, lo primero que se hace es insertar espacios (representados mediante el símbolo guión ‘-') en las secuencias de modo que ambas queden de igual largo. Los espacios se deben insertar de manera tal que permitan aumentar el grado de similitud entre ambas secuencias.
Para calcular este grado de similitud se asigna un puntaje de acuerdo a una matriz del modo que se explicará a continuación.
Una forma de alinear los genes AGTGATG y GTTAG es la siguiente:
AGTGAT-G
-GT--TAG
En este caso tenemos 4 coincidencias G en la segunda posición, T en la tercera, T en la sexta y G en la octava posición.
Otra forma de alinear ambos genes es la siguiente
AGTGATG
-GTTA-G
Acá tenemos también 4 coincidencias, G en la segunda posición, T en la tercera, A en la quinta posición y G en la octava posición.
¿Cuál alineamiento es el mejor?
Para determinarlo calculamos el puntaje de alineamiento, usando la siguiente matriz simétrica de puntajes:
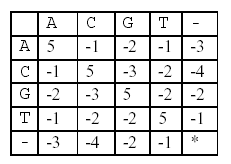
Lo que hacemos es que vemos todos los pares que se forman y sumamos los valores de acuerdo a la matriz (el asterisco indica que no pueden haber pares -,-).
En el caso del primer alineamiento tenemos los siguientes pares:
A-,GG,TT,G-,A-,TT,-A,GG
Buscamos en la matriz de acuerdo a estas “coordenadas”:
A-=(-3), GG=5, TT=5, G-=(-2), A-=(-3), TT=5, -A=(-3), GG=5
Sumamos: (-3)+5+5+(-2)+(-3)+5+(-3)+5=9
Hagamos el mismo cálculo para el segundo alineamiento:
A-=(-3), GG=5, TT=5, GT=(-2), AA=5, T-=(-1), GG=5
Sumamos: (-3)+5+5+(-2)+5+(-1)+5 = 14
En este caso el puntaje de alineamiento es 14 por lo tanto este alineamiento es mejor.
El problema es encontrar un algoritmo que dados dos genes encuentre el alineamiento con el mayor puntaje.
Desafío Marzo-Abril 2013
Se ha cometido un asesinato. Entre las evidencias se tiene ADN de la víctima y del presunto asesino. Sin embargo, después de secuenciar el ADN el servidor que contienen el software que permite comparar las muestras ha sufrido un crash de disco. El equipo forense ha sido descuidado y no tiene respaldos.
Lo lamentable es que el computador contenía la implementación de un notable algoritmo de Alineamiento de Secuencias de Genes desarrollado por un prestigioso investigador con el que se ha perdido contacto.
Por lo tanto nos han pedido que propongamos distintos algoritmos de alineamiento de secuencias y que comparemos el ADN recogido en la escena del crimen contra el genoma de varios sospechosos.
Así que el desafío de esta oportunidad consiste en construir un programa que reciba un archivo con la siguiente forma:
- En que la primeras 5 lineas viene la matriz de puntajes
- Después viene la evidencia (el ADN recogido en la escena del crimen)
- Finalmente vienen las secuencias de ADN de varios sospechosos.
- El archivo puede venir con comentarios, los que empiezan con el carácter #.
- Las lineas que comienzan con las letras A,C,G,T y el guión corresponden a los datos de la matriz, la linea que comienza con el numero 0 corresponde a la evidencia, y las lineas que comienzan con un número mayor o igual 1 corresponden a los ADN de los sospechosos.
El siguiente es un ejemplo de archivo de entrada:
# Matriz
A:5,-1,-2,-1,-3
C:-1,5,-3,-2,-4
G:-2,-3,5,-2,-2
T:-1,-2,-2,5,-1
-:-3,-4,-2,-1,*
# Evidencia
0:AGTGATG
# ADN Sospechosos
1:AAATGC
2:AGGAA
3:AGTGATA
4:GATTACA
El programa debe entregar como resultado el número del sospechoso cuyo ADN se parece más al de la evidencia.
En este caso, con el archivo de entrada mostrado anteriormente la salida del programa debe ser:
El culpable es el sospechoso número 3 (AGTGATA).
Opcionalmente el programa puede mostrar los alineamientos y el puntaje de alineamiento, pero no es necesario para participar (aunque si hay un empate, el programa con más “features” tiene más “puntos”).
Ganará el programa que se demore el menor tiempo en calcular el culpable. En caso de haber empate, o tener tiempos muy similares, se calculará el valor E de acuerdo a las métricas de Halstead.
El premio por este desafío será una Giftcard Amazon de 40 dólares la que se entregará sólo si participan 8 o más concursantes.
El código fuente de los programas debe estar disponibles en un repositorio público GitHub o Bitbucket, no se aceptarán programas entregados en otro medio.
Que empiece la competencia, y que gane el “más mejor”, el ganador se determinará el día 1 de mayo.